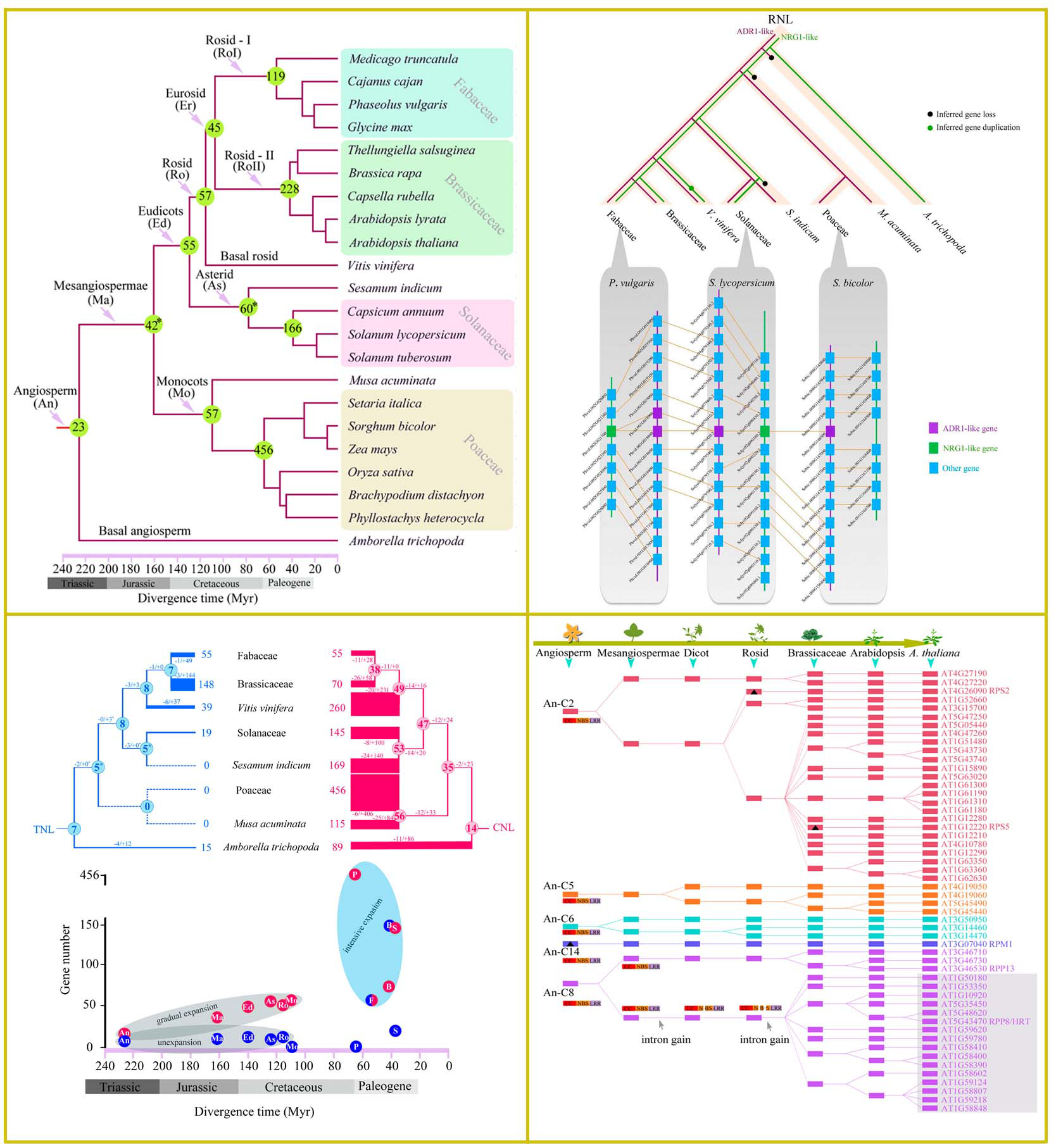
我校伟德BETVlCTOR1946陈建群教授课题组在植物抗病基因的演化研究中取得重要进展,相关研究成果“Large-scale analyses of angiosperm nucleotide-binding site-leucine-rich repeat (NBS-LRR) genes reveal three anciently diverged classes with distinct evolutionary patterns”于2016年02月02日在线发表在植物学顶级期刊之一《Plant Physiology》(SCI一区,5年平均影响因子8.0)。第一作者为我校博士后邵珠卿博士,通讯作者为陈建群教授和王斌副教授。
在该论文中,团队研究人员对植物最大的一类抗病基因――NBS-LRR基因在被子植物中的演化模式进行了研究。通过对22个被子植物基因组中鉴定出的6000多条NBS-LRR基因进行系统演化分析,提出了NBS-LRR 基因划分的新体系;从被子植物演化尺度上揭示了该类基因的演化历史、演化模式及机制。结果发现:1)NBS-LRR基因包括三个古老的亚类:TNL、CNL和RNL。作为参与抗病信号传导的基因,RNL呈现出保守的演化特征;而直接参与病原识别的TNL和CNL基因则展现出不同的扩张模式;2)在白垩纪和第三纪过渡期(K-P boundary)的真菌爆发可能是NBS-LRR基因剧烈扩张的一个重要诱因;3)对功能基因演化历程的追溯揭示了植物与病原间的长期“军备竞赛”。
该研究首次从被子植物的演化尺度上对NBS-LRR基因的演化进行了系统的研究,深入揭示了植物抗病基因的演化模式以及植物与病原的共演化历程,同时也对新的抗病基因的发掘和利用具有重要参考意义。
据悉,该研究团队2014年也在《Plant Physiology》发表论文 “Long-term evolution of nucleotide-binding site-leucine-rich repeat genes: understanding gained from and beyond the legume family”,从豆科植物的演化尺度上揭示了NBS-LRR基因在豆科物种分化过程中持续扩增的演化模式和以串联复制为主的扩张机制;并对大豆抗花叶病毒(SMV)复合抗病位点的形成和演化机制进行了深入探讨(Plant Physiology, 166(1), 217-234)。论文第一作者同样为邵珠卿博士,通讯作者为陈建群教授和王斌副教授。
该研究得到国家自然科学基金重大研究计划,国家自然科学基金面上项目、青年基金项目,中国博士后基金特别资助、一等资助,江苏省青蓝工程“创新团队”及中央高校基本科研业务费专项资金等项目的资助。
我校伟德BETVlCTOR1946陈建群教授课题组在植物抗病基因的演化研究中取得重要进展,相关研究成果“Large-scale analyses of angiosperm nucleotide-binding site-leucine-rich repeat (NBS-LRR) genes reveal three anciently diverged classes with distinct evolutionary patterns”于2016年02月02日在线发表在植物学顶级期刊之一《Plant Physiology》(SCI一区,5年平均影响因子8.0)。第一作者为我校博士后邵珠卿博士,通讯作者为陈建群教授和王斌副教授。
在该论文中,团队研究人员对植物最大的一类抗病基因――NBS-LRR基因在被子植物中的演化模式进行了研究。通过对22个被子植物基因组中鉴定出的6000多条NBS-LRR基因进行系统演化分析,提出了NBS-LRR 基因划分的新体系;从被子植物演化尺度上揭示了该类基因的演化历史、演化模式及机制。结果发现:1)NBS-LRR基因包括三个古老的亚类:TNL、CNL和RNL。作为参与抗病信号传导的基因,RNL呈现出保守的演化特征;而直接参与病原识别的TNL和CNL基因则展现出不同的扩张模式;2)在白垩纪和第三纪过渡期(K-P boundary)的真菌爆发可能是NBS-LRR基因剧烈扩张的一个重要诱因;3)对功能基因演化历程的追溯揭示了植物与病原间的长期“军备竞赛”。
该研究首次从被子植物的演化尺度上对NBS-LRR基因的演化进行了系统的研究,深入揭示了植物抗病基因的演化模式以及植物与病原的共演化历程,同时也对新的抗病基因的发掘和利用具有重要参考意义。
据悉,该研究团队2014年也在《Plant Physiology》发表论文 “Long-term evolution of nucleotide-binding site-leucine-rich repeat genes: understanding gained from and beyond the legume family”,从豆科植物的演化尺度上揭示了NBS-LRR基因在豆科物种分化过程中持续扩增的演化模式和以串联复制为主的扩张机制;并对大豆抗花叶病毒(SMV)复合抗病位点的形成和演化机制进行了深入探讨(Plant Physiology, 166(1), 217-234)。论文第一作者同样为邵珠卿博士,通讯作者为陈建群教授和王斌副教授。
该研究得到国家自然科学基金重大研究计划,国家自然科学基金面上项目、青年基金项目,中国博士后基金特别资助、一等资助,江苏省青蓝工程“创新团队”及中央高校基本科研业务费专项资金等项目的资助。
我校伟德BETVlCTOR1946陈建群教授课题组在植物抗病基因的演化研究中取得重要进展,相关研究成果“Large-scale analyses of angiosperm nucleotide-binding site-leucine-rich repeat (NBS-LRR) genes reveal three anciently diverged classes with distinct evolutionary patterns”于2016年02月02日在线发表在植物学顶级期刊之一《Plant Physiology》(SCI一区,5年平均影响因子8.0)。第一作者为我校博士后邵珠卿博士,通讯作者为陈建群教授和王斌副教授。
在该论文中,团队研究人员对植物最大的一类抗病基因――NBS-LRR基因在被子植物中的演化模式进行了研究。通过对22个被子植物基因组中鉴定出的6000多条NBS-LRR基因进行系统演化分析,提出了NBS-LRR 基因划分的新体系;从被子植物演化尺度上揭示了该类基因的演化历史、演化模式及机制。结果发现:1)NBS-LRR基因包括三个古老的亚类:TNL、CNL和RNL。作为参与抗病信号传导的基因,RNL呈现出保守的演化特征;而直接参与病原识别的TNL和CNL基因则展现出不同的扩张模式;2)在白垩纪和第三纪过渡期(K-P boundary)的真菌爆发可能是NBS-LRR基因剧烈扩张的一个重要诱因;3)对功能基因演化历程的追溯揭示了植物与病原间的长期“军备竞赛”。
该研究首次从被子植物的演化尺度上对NBS-LRR基因的演化进行了系统的研究,深入揭示了植物抗病基因的演化模式以及植物与病原的共演化历程,同时也对新的抗病基因的发掘和利用具有重要参考意义。
据悉,该研究团队2014年也在《Plant Physiology》发表论文 “Long-term evolution of nucleotide-binding site-leucine-rich repeat genes: understanding gained from and beyond the legume family”,从豆科植物的演化尺度上揭示了NBS-LRR基因在豆科物种分化过程中持续扩增的演化模式和以串联复制为主的扩张机制;并对大豆抗花叶病毒(SMV)复合抗病位点的形成和演化机制进行了深入探讨(Plant Physiology, 166(1), 217-234)。论文第一作者同样为邵珠卿博士,通讯作者为陈建群教授和王斌副教授。
该研究得到国家自然科学基金重大研究计划,国家自然科学基金面上项目、青年基金项目,中国博士后基金特别资助、一等资助,江苏省青蓝工程“创新团队”及中央高校基本科研业务费专项资金等项目的资助。
2016文章链接:
http://www.plantphysiol.org/content/early/2016/02/02/pp.15.01487.abstract
2014文章链接:
http://www.plantphysiol.org/content/166/1/217.long